
Détails du challengeDéroulement du challenge
Le challenge se déroulera en trois phrases :
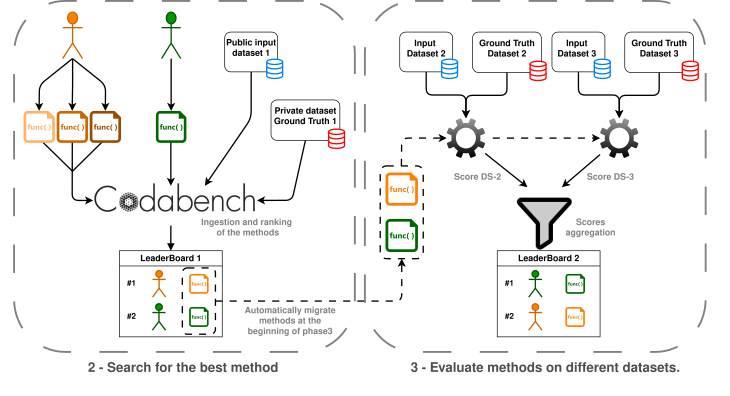
Et en pratique, comment ça marche ?Les participant⋅e⋅s travailleront par équipe de 4 personnes. Chacun⋅e travaillera sur son ordinateur portable personnel et aura au préalable installé les environnements de développement nécessaires au challenge (Docker, R et packages associés). 1. Chaque équipe développe des solutions en local, sur ses ordinateurs personnels. 2. Chaque équipe soumet ses solutions (son code et/ou les résultats) sur la plateforme du challenge, Codabench. 3. Le code est exécuté sur la plateforme en utilisant un environnement Docker standardisé. Si des résultats sont soumis, ceux-ci sont comparés avec les résultats présents sur la plateforme (inconnus des équipes). 4. Il sera possible de faire plusieurs soumissions par équipe. Les performances de chaque soumission seront évaluées. 5. La meilleure solution proposée (développée en R) de chaque équipe sera ensuite transférée pour l'évaluation finale de la phase 3 du challenge. 6. L'image Docker sera mise à disposition des participant⋅e⋅s afin que toutes les équipes puissent travailler dans un environnement standardisé (afin d'éviter au maximum les problèmes de compatibilité). En résumé, chaque équipe développera sa solution au problème localement, puis la soumettra sur Codabench. L'exécution et l'évaluation de cette solution sera réalisée via un environnement Docker standardisé. Plateforme de soumission : CodabenchLe challenge est hébergé sur Codabench, plateforme de data challenges : https:// Les équipes soumettront leurs solutions sur cette plateforme. Les solutions y seront éxécutées et évaluées via une image Docker. Le lien du challenge sera prochainement communiqué. Quésaco la déconvolutionLa déconvolution de données bulk RNAseq permet d'estimer la proportion des différentes populations cellulaires présentes dans un échantillon à partir de leur profils d'expression génique. Cette information peut être utilisée pour étudier l'hétérogénéité cellulaires des tumeurs.
Données du challenge
Différents jeux de données seront utilisés lors de ce challenge :
Références[1] Meichen Dong, Aatish Thennavan, Eugene Urrutia, Yun Li, Charles M Perou, Fei Zou, Yuchao Jiang, SCDC: bulk gene expression deconvolution by multiple single-cell RNA sequencing references, Briefings in Bioinformatics, Volume 22, Issue 1, January 2021, Pages 416–427, https://doi.org/10.1093/bib/bbz166 |

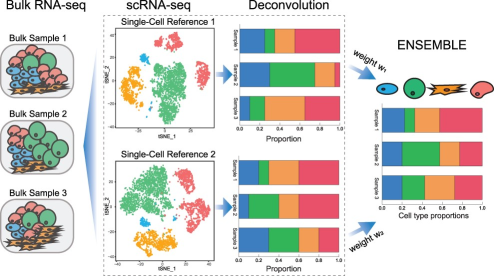 Aperçu schématique d'une déconvolution de données bulk RNAseq en utilisant une référence single-cell RNAseq. Cette figure est la Figure 1 du papier [1].
Aperçu schématique d'une déconvolution de données bulk RNAseq en utilisant une référence single-cell RNAseq. Cette figure est la Figure 1 du papier [1]. 
